无细胞蛋白表达
选择细胞裂解物对活细胞中重组蛋白表达是很有用的,它们可以裂解真核细胞或细菌细胞,并去除蛋白质中所有不需要表达的成分。这些裂解物可以与DNA模板和其他所需成分在试管中结合,进行蛋白质的表达(1)。
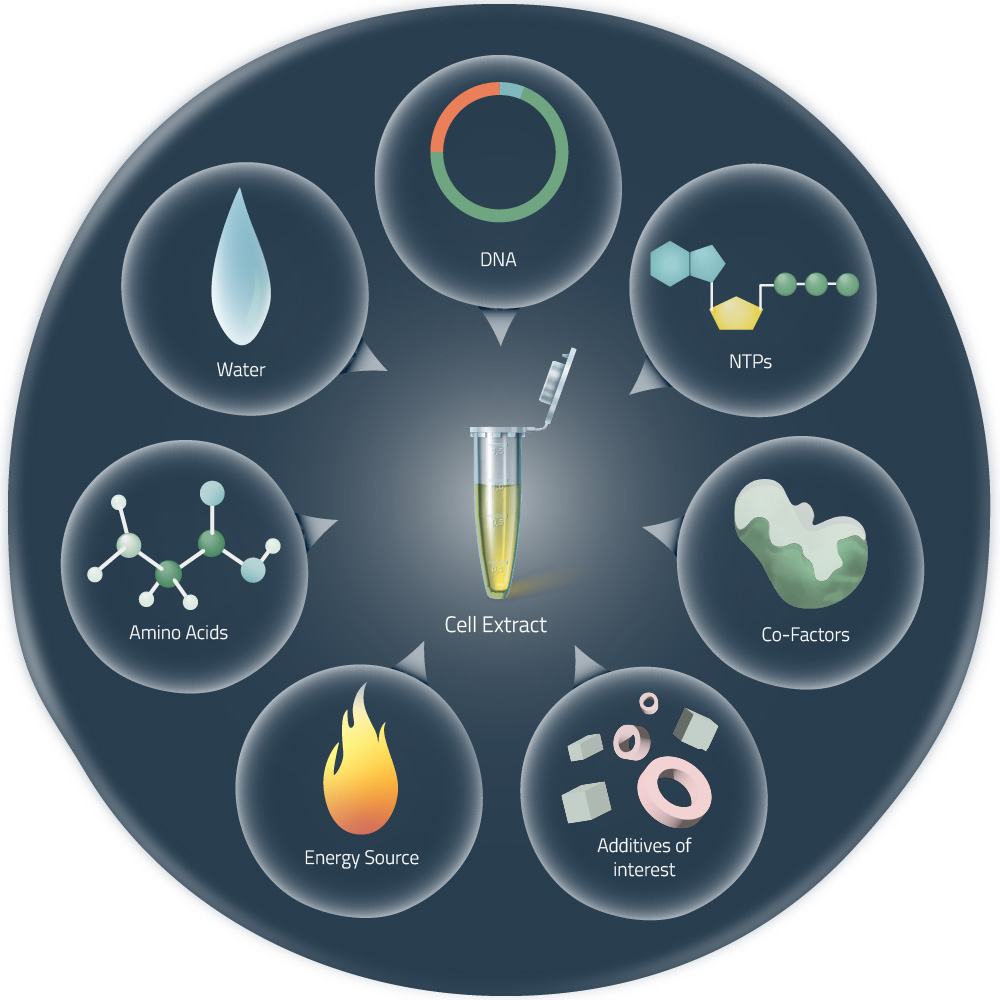
Figure 1: Cell-free expression components and additives
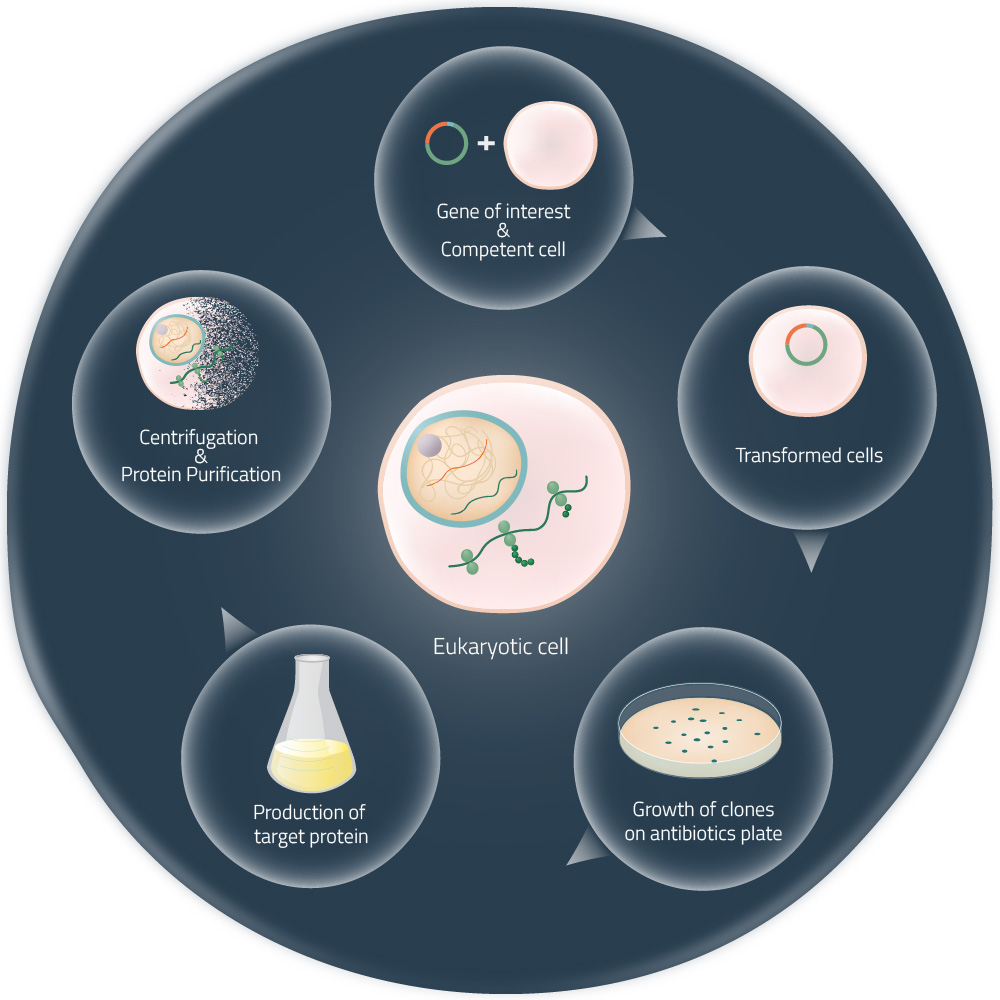
Figure 2: Production of a target protein with living cells
优势
1. 无细胞反应很容易建立,仅需几个小时就可以完成蛋白质表达。这种方法可以用小规模(50-100微升)的反应快速完成反应条件的优化,优秀的条件能被线性放大到其所需的数量。
2. 总处理量小:对于可溶性蛋白,几毫升的反应就可以得到毫克量的蛋白。
3. 作为一个开放的系统,许多成分(天然的和非天然的),均可以被添加到反应混合物中,如还原/氧化元素、分子伴侣、标记的氨基酸或去污剂。如此,蛋白质可以直接用于核磁共振,也可将膜蛋白在新生状态下重组到纳米盘系统中,而无需任何人造去污剂(10-12)。
4. 真核细胞的裂解物能进行翻译后修饰,如糖基化或磷酸化,并可将天然的细胞膜成分(如微粒体)插入膜蛋白(9)。
5. 通常可以在无细胞系统中进行活细胞毒性蛋白的表达,由于已经去除了负责(功能)的细胞成分,所以不会受到如蛋白消化酶或DNA模板不稳定的影响。
6. 多聚体蛋白和复合物都能被表达,且DNA模板滴定会影响不同亚基的比例。
劣势
1. 如果蛋白质需求量大或者蛋白质表达低,无细胞表达会相当昂贵。因此,对于每一种蛋白质,应在小规模实验中评估可达到的产量,并计算放大的成本,这对来自真核生物更复杂的无细胞裂解物来说更应如此。
表1 不同无细胞表达系统的比较
| 大肠杆菌 | 小麦胚芽 | 昆虫 | 哺乳动物 |
相对产量 | 高(几毫克/毫升) | 中 | 中低 | 低 |
相对成本 | 低 | 中 | 中 | 高 |
磷酸化 | 无 | 有 | 有 | 有 |
糖基化 | 无 | 核心 | 核心 | 哺乳动物 |
膜蛋白选择 | 不溶性纳米磷脂盘去污剂 | 脂质去污剂 | 天然内体纳米磷脂盘 | 天然微粒体脂质去污剂 |
市售 | mastermix CECF 试剂盒批次单独的细胞裂解物 | CECF 试剂盒批次 | mastermix 批次 | mastermix 批次 |
库柏生物供应 | 大肠杆菌细胞裂解物和CECF纳米磷脂盘去污剂 |
|
|
|
应用
由于蛋白质是在一个开放性的系统中表达的,与活细胞表达相比,无细胞表达使许多应用受益,包括:
·表达筛选:通过无细胞表达,您可以在两天内从DNA模板-甚至是多个平行应得到免疫印迹的结果,这使得该系统对于快速筛选多个构建体和反应条件更具吸引力。
·蛋白标记:开放式系统可以与标记的氨基酸相结合,例如生物素或荧光标签。
·核磁共振:可以很容易地将同位素标记的氨基酸添加到混合物中,获得准备用于核磁共振的蛋白质。
·结晶:与硒代蛋氨酸结合,在一个简单的步骤中获得蛋白质重金属衍生物。
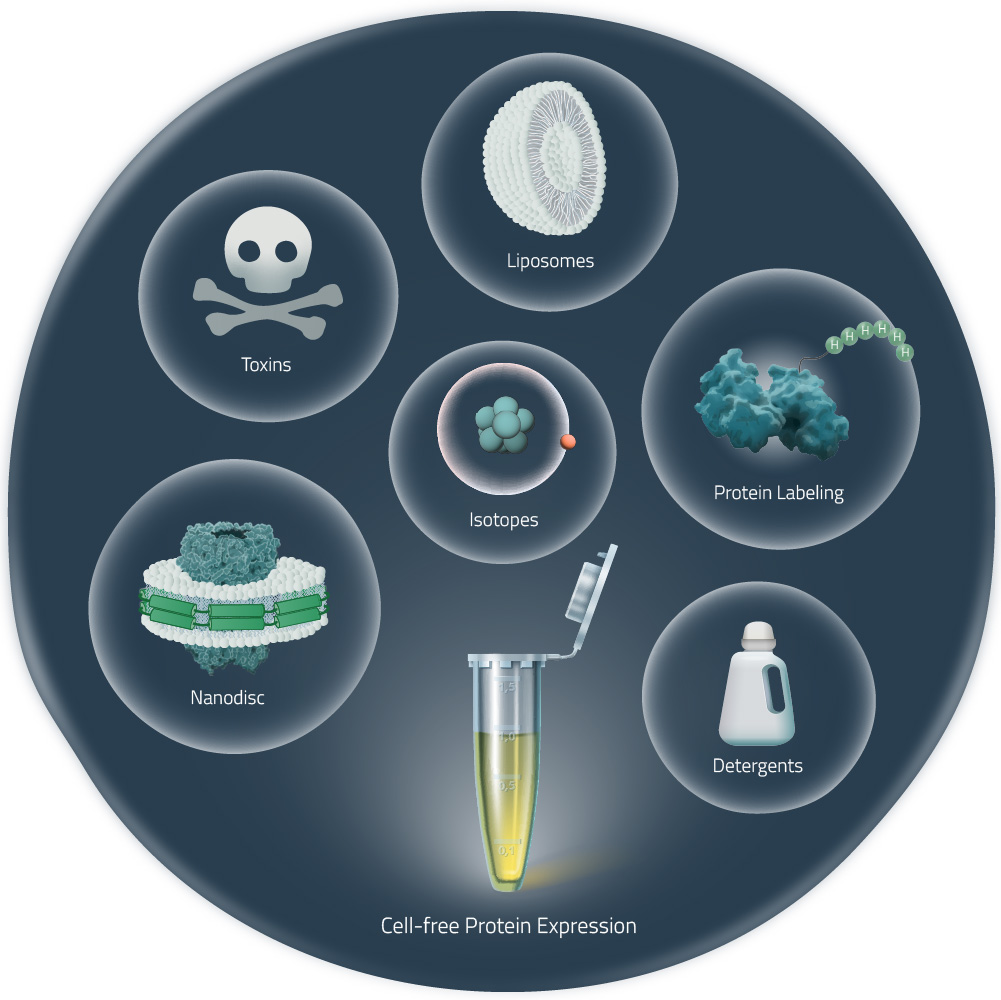
Figure 2: Production of a target protein with living cells
膜蛋白特殊的适应性
由于蛋白质是在一个开放的系统中表达的,与活细胞表达相比,无细胞表达促进了许多应用,包括:
·不溶性表达:在不添加任何种类的脂质或去污剂的情况下,形成膜蛋白聚集体,在诸多情况下可以重新折叠成功能蛋白。
·去污剂:尤其是Brij系列的去污剂,多数情况下将其加入以溶解膜蛋白。如果需要,可以在后续步骤中交换去污剂,但需要是在纯化过程中。(2-7)
·纳米磷脂盘:在无细胞反应中加入纳米磷脂盘,是获得稳定性蛋白质简单和简便的方法,不需要去污剂,可用于各种检测(10-15)。其应用范围包括生物物理学分析、表面等离子共振、核磁共振和冷冻电镜的质谱分析。最近有研究表明,通过纳米磷脂盘,膜蛋白在无细胞裂解物中的表达可通过中观方法进行结晶(14),了解更多关于纳米磷脂盘的信息。
·脂质体和其他脂质颗粒:如脂质体、微粒体和其他含脂质的结构(如双层膜微胞),已被添加到无细胞反应中,尤其是与昆虫细胞裂解物结合进行膜蛋白的表达(9)。使用适量的去污剂,还可以将膜蛋白从纳米磷脂盘转移到双层膜微胞中(13)。
间歇式和无细胞持续交换方法
通常可按两种方法进行无细胞反应:
1. 间歇式:可以轻松的在简易管中进行间歇式无细胞反应,如离心管。其反应时间很短(1-3小时),因为能量物质很快就被消耗完了,而最终产物的积累会减缓酶促反应的速度,因此蛋白质的产量通常会很低。
2.无细胞持续交换或半透膜:无细胞持续交换(CECF)反应是通过半透膜进行的,所以可向反应提供能量物质,并去除最终产物,如ADP。如此,反应时间就可以达到24小时,对于表达量高的可溶性蛋白质,蛋白质每毫升的产量可以达到几毫克(8)。
References:
1. Endo, Y. et al. (eds) Cell-Free Protein Production, Methods and Protocols. Methods in Molecular Biology vol. 607 (2010)
2. Nirenberg, M. W., Matthaei, J. H. "The Dependence of Cell- Free Protein Synthesis in E. Coli Upon Naturally Occurring or Synthetic Polyribonucleotides". Proceedings of the National Academy of Sciences of the United States of America. 47 (10): 1588–1602 (1961)
3. Roos, C. et al. Characterization of co-translationally formed nanodisc complexes with small multidrug transporters, proteorhodopsin and with the E.coli MraY translocase. Biochim. Biophys. Acta 1818; 3098-3106 (2012)
4. Proverbio, D. et al. Functional properties of cell-free expressed human endothelin A and endothelin B receptors in artificial membrane environments. Biochim. Biophys. Acta 1828, 2182-92 (2013)
5. Roos, C. et al. High-level cell-free production of membrane proteins with nanodiscs. In: Cell-free Protein Synthesis: Methods and Protocols. Alexandrov, K., and Johnston, W.A. (eds), Methods in Molecular Biology vol. 1118, Chapter 7 (2014)
6. Sachse, R. et al. Membrane protein synthesis in cell-free systems: From bio-mimetic systems to bio-membranes. FEBS Lett. 588,2774-2781 (2014)
7. Jackson, A.M. "Cell-free protein synthesis for proteomics". Briefings in Functional Genomics and Proteomics. 2 (4): 308–319 (2004)
8. Court, R., Cook, N., Saikrishnan, K., Wigley, D. The Crystal Structure of λ-Gam Protein Suggests a Model for RecBCD Inhibition, Journal of Molecular Biology, Volume 371, Issue 1 (2007)
9. Suzuki, T. et al. An insect cell-free system for recombinant protein expression using cDNA resources. Methods in molecular biology vol. 577 (2009): 97-108.
10. Boland, C. et al. Cell-free expression an in meso crystallization of an integral membrane kinase for structure determination. Cell. Mol. Life Sci (2014)
11. Hein, C. et al. Hydrophobic environments in cell-free systems: Designing artificial environments for membrane proteins. J. Eng. Life Sci. 14, 365-79 (2014)
12. Proverbio, D. et al. Membrane protein quality control in cell-free expression sysems: Tools, strategies and case studies. Membrane proteins production for structural analysis (Isabelle Mus-Veteau, ed.) Springer Heidelberg. ISBN:978-1-4939-0662-8.
13. Haberstock, S. et al. A systematic approach to increase the efficiency of membrane protein production in cell-free expression systems. Prot. Exp. Purific. 82, 308-16 (2012)
14. Matthies, D. et al. Cell-free expression and assembly of a macromolecular membrane protein complex. J. Mol. Biol. 413, 593-603 (2011)
15. Schneider, B. et al. Membrane protein expression in cell-free systems. Metho. Mol. Biol. 601, 165-86 (2010)
16. Laguerre, A. et al. From nanodiscs to isotropic bicelles: A procedure for solution nuclear magnetic resonance studies of detergent-sensitive integral membrane proteins. Structure 24, 1-12 (2016)
17. Nikolaev, M. et al. Integral membrane proteins can be crystallized directly from nanodiscs. Cryst. Growth Des. 17(3), 945–948 (2017)
18. Henrich, E. et al. Analyzing native membrane protein assembly in nanodiscs by combined non-covalent mass spectrometry and synthetic biology. eLife 6:e20954 (2017)
19. Schwarz, D. et al. Preparative scale expression of membrane proteins in E.coli based continuous exchange cell-free systems. Nat. Protocols 2, 2945-57 (2007)










 Home
Home Facebook
Facebook WeChat
WeChat Twiitter
Twiitter
