如何利用GST标签蛋白纯化蛋白(下)
如何在质粒中添加/克隆GST标签
将GST标签添加到目标蛋白的优先方法是使用pGEX系列载体,它们有不同的蛋白质裂解位点,而这对于后续提到的去除GST标记方法尤其重要。图4描述了我们推荐克隆方法的过程和步骤。
(1)pGEX载体都有一个包含多个限制性酶切位点的多克隆位点(MSC),这些位点因pGEX载体而异。如表2所述。此外,所有的pGEX载体均含有三个终止密码子,覆盖了MSC之后DNA菌株上所有的三个可能的阅读框,以确保所表达的蛋白质本身并不包含终止密码子。

图4:在目标蛋白质上添加GST标签的克隆过程概述
(2)下一步是得到一个目的基因/蛋白质的PCR产物,该产物在两个方向上均带有所选pGEX载体MCS中存在的限制性酶切位点。这些引物应满足以下条件:1. 3-4 nts的悬臂(越长越有利于限制性酶切反应,建议3-4个)
2. 限制性位点
3. 20-25 nts的目标基因
以在eGFP上添加一个GST标签为例
图例 – 上游引物悬臂
限制性位点 -> 以BamHI为例
eGFP的前20 nts5'-GGG GGATCC ATGAAACATCACCATCACCA-3'
图例 –下游引物悬臂
限制性位点 -> 以EcoRI为例,反向互补
eGFP的最后20 nts,反向互补
5'-GGG GGATTC TTTGTATAGTTCATCCATGC-3'
注意本文仅是介绍如何将GST标签添加目的蛋白质上,其他方法如酵母或细菌的同源重组也可完成。
表2: 不同的pGEX载体和其MCS内的限制性酶切位点从5'到3'端排序
pGEX 载体 | 限制性酶切位点从5'到3'端排序 |
pGEX-1λT | BamHI , EcoRI |
pGEX-2T | BamHI, SmaI, EcoRI |
pGEX-2TK | BamHI, SmaI, EcoRI |
pGEX-4T-1 | BamHI, EcoRI, SmaI, SalI, XhoI, NotI |
pGEX-4T-2 | BamHI, EcoRI, SmaI, SalI, XhoI, NotI |
pGEX-4T-3 | BamHI, EcoRI, SmaI, SalI, XhoI, NotI |
pGEX-3X | BamHI, SmaI, EcoRI, |
pGEX-5X-1 | BamHI, EcoRI, SmaI, SalI, XhoI, NotI |
pGEX-5X-2 | BamHI, EcoRI, SmaI, SalI, XhoI, NotI |
pGEX-5X-3 | BamHI, EcoRI, SmaI, SalI, XhoI, NotI |
pGEX-6P-1 | BamHI, EcoRI, SmaI SalI, XhoI, NotI |
pGEX-6P-2 | BamHI, EcoRI, SmaI SalI, XhoI, NotI |
pGEX-6P-3 | BamHI, EcoRI, SmaI SalI, XhoI, NotI |
GST标签蛋白的纯化是如何进行的?
GST标签蛋白纯化是一种亲和层析法,它利用GST对还原谷胱甘肽(其天然底物)的高亲和力,依据所优选的纯化方法将谷胱甘肽与琼脂糖凝胶或磁珠结合而达到纯化的目的。如图5为GST-标签亲和微球的示意图。不同供应商的GST标签亲和产品的实际纯化方案是不同的,包括仅针对其自身特定的GST亲和力珠进行优化的不同缓冲液成分间的差异。因此,建议不要将一个供应商的GST标签蛋白纯化方案应用于其他供应商产品。
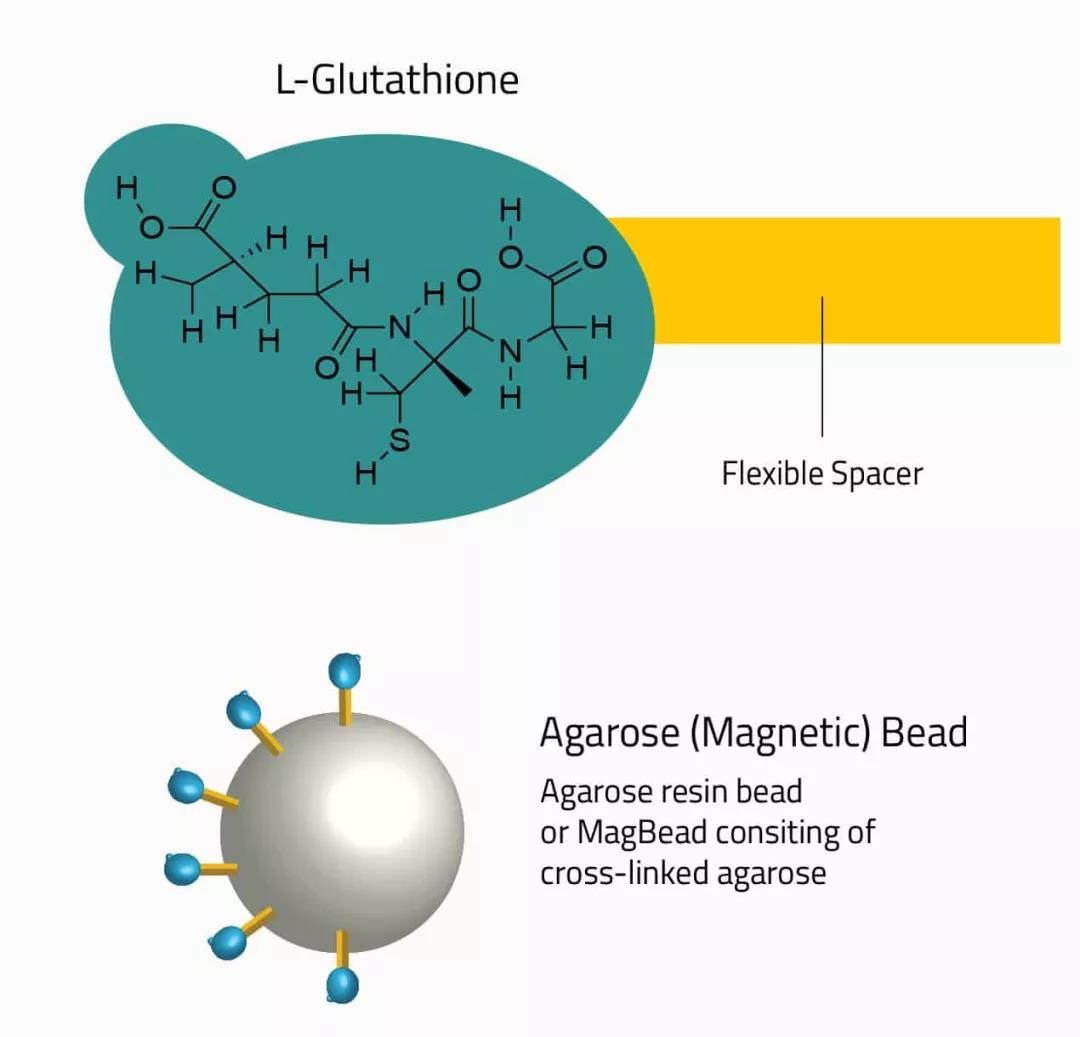
图5:GST-标签亲和微球的示意图。通过微球表面的柔性间隔物将谷胱甘肽三肽与微球相结合,也可用于与GST标签蛋白的结合。
GST标记的蛋白质纯化方案
图6介绍了用GST亲和磁珠进行蛋白质纯化的过程,但这些核心步骤仅基于使用GST-标签亲和磁珠进行纯化。
步骤1:加入细胞裂解液后细胞裂解,细胞的整个蛋白质组悬浮在裂解缓冲液内,但带有GST标签的蛋白未与其它蛋白质分离。
步骤2:在细胞裂解液中加入GST亲和标签的磁珠,加入的磁珠量应多于细胞裂解液中带GST标签目标蛋白的数量。
步骤3:旋转管/烧瓶几个小时,使谷胱甘肽磁珠与细胞裂解液内的所有GST标签蛋白充分结合。
步骤4:由于GST标签蛋白与谷胱甘肽磁珠的结合特异性,所以仅带有GST标签的目标蛋白会与磁珠结合,而不会与其它蛋白质结合。
步骤5:对细胞裂解液施加磁力,使GST标签蛋白与谷胱甘肽珠结合,而不会含有其它蛋白质。
步骤6:去除未结合的细胞裂解液/蛋白质上清, 带有GST标签的目标蛋白现已被纯化。
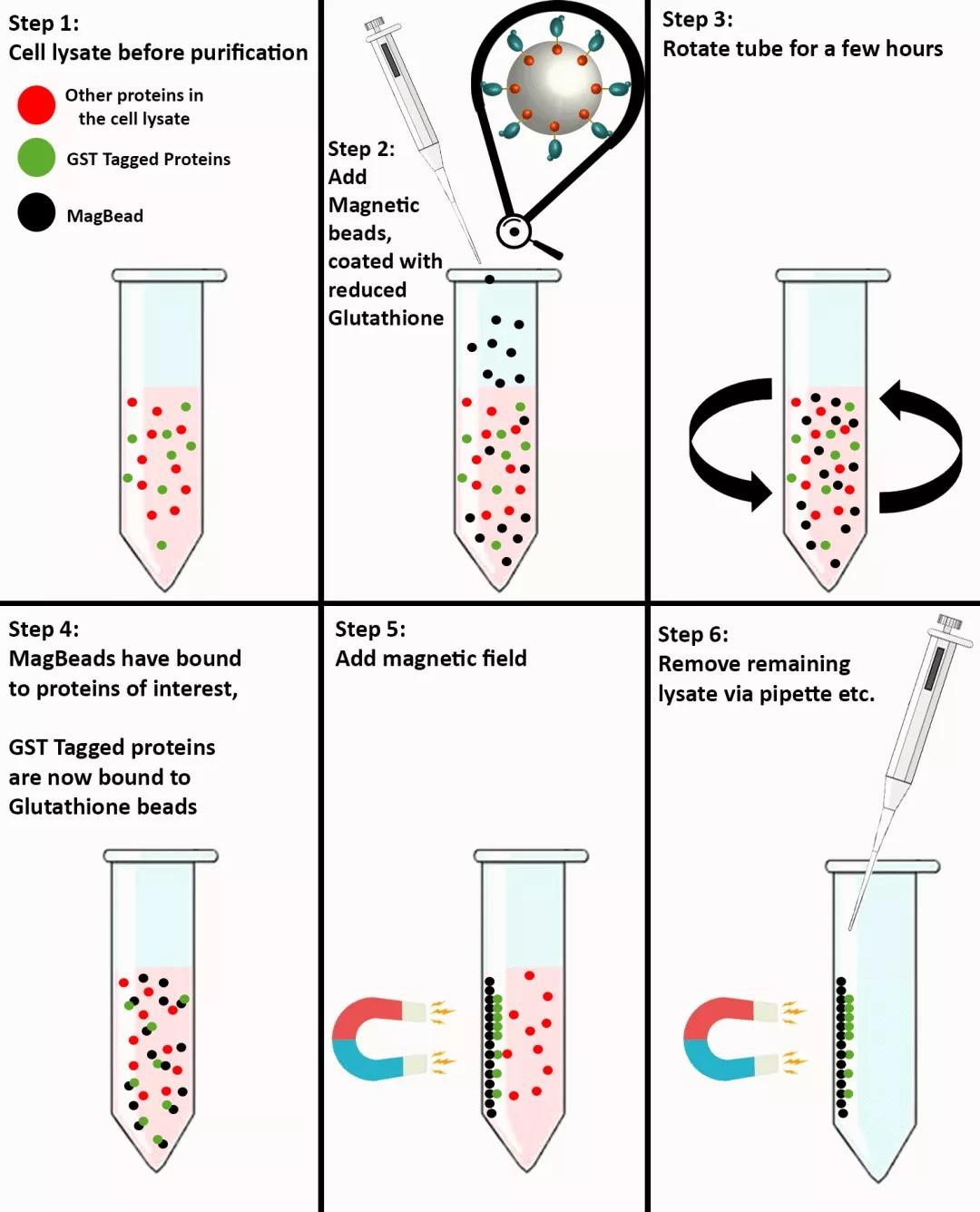
图6:使用磁珠纯化带有GST标签的目标蛋白的示例性工作流程概述。
为了验证带有GST标签的目标蛋白是否纯化成功,建议利用Western Blot(蛋白质印迹法)通过抗GST抗体检测纯化蛋白。蛋白质印迹法示例可在此查看。
如何去除GST标签?
GST标签有26千道尔顿(kDa),是目前最大的亲和标签之一。作为一个完整的蛋白质,它甚至会大于它所结合的一些蛋白质,因此,科研者就会担忧,这种大小的蛋白质人工连接在另一个蛋白质上是否会干扰蛋白质的功能性。所以是否有可能在蛋白质纯化后去除GST标签,以确保它不会以任何方式阻碍纯化了的蛋白质后续研究。pGEX载体系列的创建者已经考虑到了这个问题,所以在表2中也已经提到。按照“如何在质粒中添加/克隆GST标签 ”一章中描述的方式创建GST标签融合蛋白后可以选择在其后去除此GST标签。在质粒的MCS的5'端添加一个特定的蛋白裂解位点,即使在纯化后,N-末端的GST-标记也可以被裂解,这也可以作为纯化过程中的一种洗脱方法,而不仅仅是在琼脂糖树脂和磁珠纯化方案中提到的洗脱缓冲液。由于这种方法可能会有小概率使目标蛋白质包含对应的蛋白酶切位点,所以在pGEX载体中有三种不同的蛋白裂解位点可供选择。
表3:不同的pGEX载体及其包括的蛋白酶裂解位点。
pGEX载体 | MCS的5'端的蛋白酶切位点 |
pGEX-1λT; pGEX-2T; pGEX-2TK; pGEX-4T-1; pGEX-4T-2; pGEX-4T-3 | Thrombin |
pGEX-3X; pGEX-5X-1; pGEX-5X-2; pGEX-5X-3 | Factor Xa |
pGEX-6P-1; pGEX-6P-2; pGEX-6P-3 | PreScission Protease |
GST标签去除方案(示例)
注意:该方案描述了如何使用凝血酶裂解位点从自由洗脱的GST融合蛋白上裂解GST标签。
表4:凝血酶裂解所需的试剂
试剂 | 剂量 |
Thrombin | 取决于供应商:通常3-10U(单位)的凝血酶(Thrombin )可以在室温过夜条件下裂解1mg融合蛋白。详情请参见供应商说明书 |
1 X PBS buffer | 140mM NaCl; 2.7mM KCl; 10mM Na2HPO4; 1.8mM KH2PO4; pH 7.3 |
PMSF | 准备至少1mM适用于您的蛋白上清浓度 |
AEBSF | 也可以使用PMFS;准备至少1mM适用于您的蛋白上清浓度 |
步骤(常规情况)
1. 在洗脱液或纯化柱中加入10个凝血酶裂解单位~10µl每毫克的GST融合蛋白
2. 轻轻混匀,将混合物在室温(22℃)下孵育2-16小时。
3. 孵育后,加入1mM的PMSF或ABESF终止反应。
4. 通过SDS检查结果。
5. 通过SDS色谱法直接分离裂解产物(如:排组层析法)。
如果已经找到了理想的裂解条件,则可以用线性的方式扩增程序。
提示:可培养足量的GST标签蛋白,并可在SDS-Page凝胶上观察到0.01至0.06单位和不同孵育时间(例如2h,4h,6h等)的凝血酶。按所述方法停止反应,并将试剂分装,放置在-20℃保存。
参考文献
1. Hayes, J. D., Flanagan, J. U. & Jowsey, I. R. GLUTATHIONE TRANSFERASES. Annu. Rev. Pharmacol. Toxicol. 45, 51–88 (2004).
2. Allocati, N., Federici, L., Masulli, M. & Di Ilio, C. Glutathione transferases in bacteria. The FEBS Journal 276, 58–75 (2009).
3. Oakley, A. Glutathione transferases: A structural perspective. Drug metabolism reviews 43, 138–51 (2011).
4. Smith, D. B. et al. Mr 26,000 antigen of Schistosoma japonicum recognized by resistant WEHI 129/J mice is a parasite glutathione S-transferase. Proc Natl Acad Sci U S A 83, 8703–8707 (1986).
5. Terpe, K. Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems. Applied Microbiology and Biotechnology 60, 523–533 (2003).
6. Rudert, F. et al. pLEF, a novel vector for expression of glutathione S-transferase fusion proteins in mammalian cells. Gene 169, 281–282 (1996).
7. Harper, S. & Speicher, D. W. Purification of proteins fused to glutathione S-transferase. Methods Mol Biol 681, 259–280 (2011).
8. Smith, D. B. & Johnson, K. S. Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase. Gene 67, 31–40 (1988).
Lu, Q., Bauer, J. C. & Greener, A. Using Schizosaccharomyces pombe as a host for expression and purification of eukaryotic proteins. Gene 200, 135–144 (1997).










 Home
Home Facebook
Facebook WeChat
WeChat Twiitter
Twiitter
